搜索


近日,6165cc金沙总站代谢与发育科学国际合作联合实验室魏冬青课题组在人工智能辅助药物发现的计算方法领域取得重要进展,并筛选出了一个针对新冠病毒的有效多肽抑制剂——人肠防御素5(HD5)。相关成果分别发表在 Briefings in Bioinformatics(https://doi.org/10.1093/bib/bbaa205)和 Gastroenterology(https://doi.org/10.1053/j.gastro.2020.05.015)。
药物发现是识别具有潜在治疗作用的新候选化合物的过程,药物-靶标相互作用(DTI)的识别和预测可以帮助理解药物作用机制,辅助新药发现或将现有药物用于新的疾病类型或靶标。实验方法鉴定DTI既耗时又耗费资源,通过计算方法预测DTI将大幅提升研究效率。在过去的十多年中,已有许多用于预测DTI的计算方法,但仍存在精度低、假阳性高等问题。为此,魏冬青课题组长期钻研于生物信息学、人工智能辅助药物设计等领域的方法学研究。前期研究工作中,该课题组开发了基于网络标签空间划分方法,将一个多标签多分类问题转化为多个单标签多分类器的集成算法,该算法考虑了类标签之间的相关性,优于传统的多分类学习算法,并首次应用于药物代谢领域(https://doi.org/10.1021/acs.jcim.9b00749)。另一方面,成功发展了深度级联森林的人工智能方法(DTI-CDF),可在小样本量数据的情况下获得性能媲美甚至优于深度学习的结果(https://doi.org/10.1093/bib/bbaa104 )。
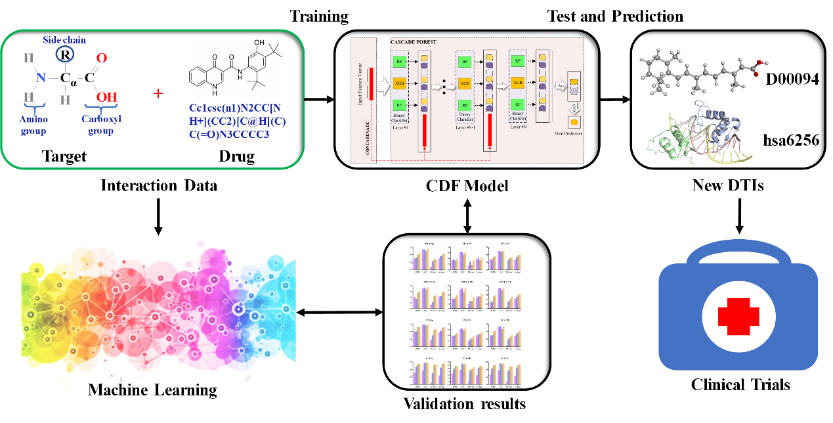
图 DTI-CDF框架
近日,在DTI-CDF方法研究的基础上,魏冬青教授与加拿大皇家学会院士、美国AAAS院士、加拿大卡尔加里大学Dennis Russell Salahub教授共同合作,进一步提出了多标签学习与社区检测方法(DTI-MLCD)。该研究中提到的基于网络标签空间划分的多标签多分类学习方法为DTI预测问题提供了一种新思路,同时为下游新药发现、老药新用等提供理论和实践依据。此外,该课题组还更新了自2008年以来由日本京都大学Yoshihiro Yamanishi教授创建并被业界同行广泛使用的金标准数据集,将DTI数量从5127增加到20025,数据量增幅了3倍,同时公开了数据集更新代码,与科研人员实时共享。
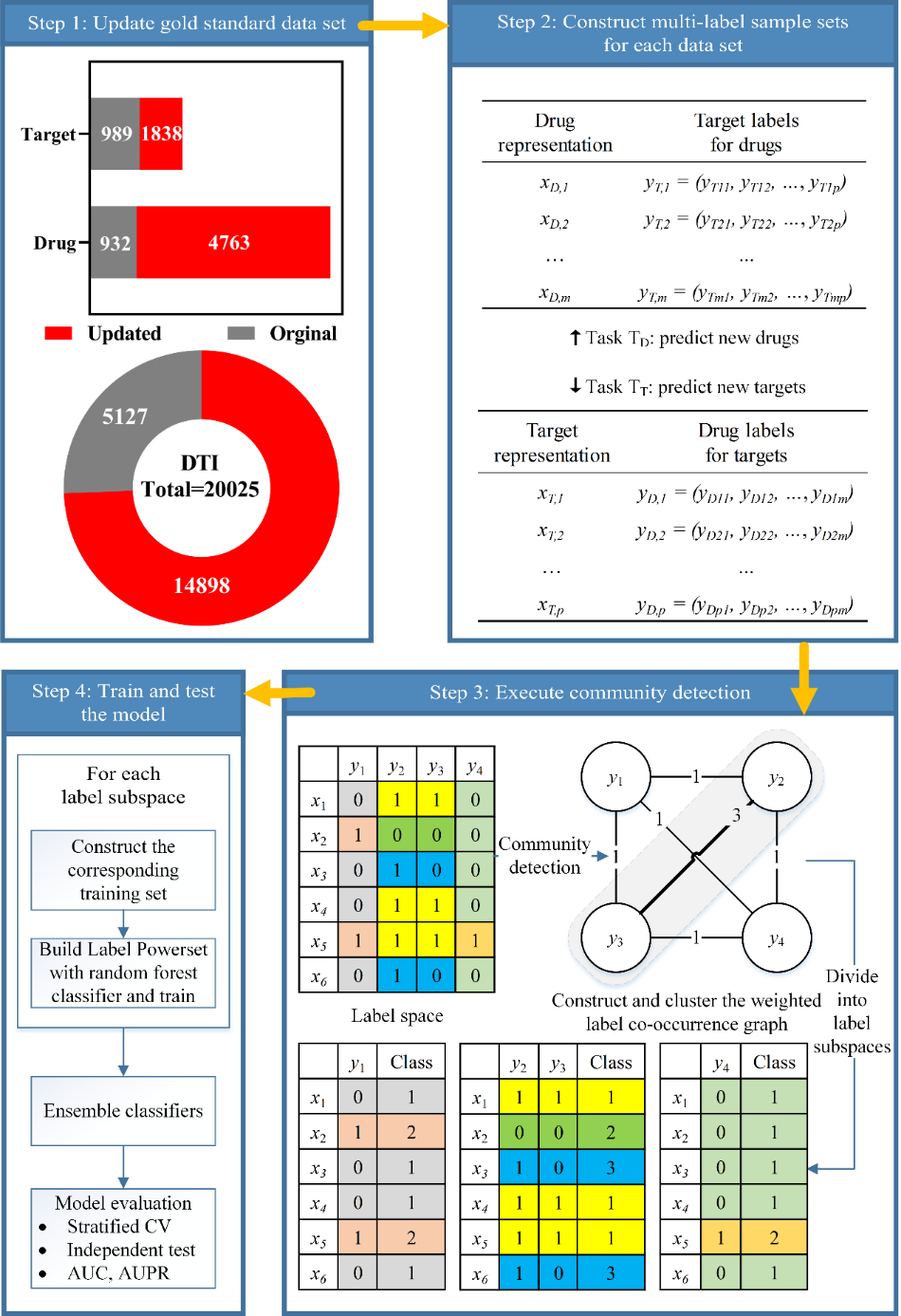
图 DTI-MLCD框架
在前期研究的基础上,魏冬青教授与上海理工大学李代禧教授合作建立了人工智能药物筛选平台,并从海量候选多肽中筛选出了多个潜在的新冠病毒有效多肽抑制剂。这些多肽抑制剂有望通过结合人体宿主细胞上的血管紧张素转化酶2,抑制新冠病毒(SARS-CoV-2)突刺蛋白对人黏膜细胞的侵袭,从而阻断新冠病毒传染途径。人工智能药物筛选平台为助力新药发现、筛选及设计提供了有力的技术支持,也为辅助理解药物作用机理提供了新的技术手段。其中,他们筛选获得的一种新冠病毒的有效中药成分——人肠防御素5(HD5)的抑制效果首先得到了合作者陆军医科大学教授王军平、赵景宏的实验验证。该成果对后续抗新冠病毒安全疫苗的研发和设计具有重要的理论价值和现实指导意义。
上述研究工作中,参与人员包括6165cc金沙总站博士生褚晏伊、代谢与发育科学国际合作联合实验室副研究员熊毅。该系列研究获得国家重点研发计划(2016YFA0501703)、国家自然科学基金重点项目(61832019)、面上项目(61872094)、青年项目(31601074)、代谢与发育科学国际合作联合实验室开放课题(MDS-JF-2019A08)、微生物代谢国家重点实验室重点课题等资助。


 首页
首页

 学院概况
学院概况

 师资队伍
师资队伍

 人才培养
人才培养

 招生就业
招生就业

 科学研究
科学研究

 平台基地
平台基地

 党群工作
党群工作

 校友之家
校友之家

 安全工作
安全工作

 网上办事
网上办事

 当前位置:
当前位置: